Hodowanie zarazków na szalce Petriego jest całkiem proste – przetrzyj wszystko wacikiem, zostaw w ciepłym pomieszczeniu na kilka dni i gotowe! Masz nowych futrzanych przyjaciół.
Ale rodzaje drobnoustrojów, które można hodować na szalce Petriego, to tylko niewielki ułamek bakterii, archeonów i innych mikroorganizmów, które osiedliły się na wymazie – tylko te, które są odpowiednie do warunków, w których je wyhodowałeś.
Ogromna większość z nich nie lubi środowiska, które możemy zapewnić i dlatego nie będą posłusznie rosnąć na szalce Petriego.
Obecnie międzynarodowy zespół naukowców odkrył 12 556 nowych gatunków bakterii i archeonów, które nigdy nie były hodowane w laboratorium przy użyciu nowej techniki zwanej metagenomiką.
„Udało nam się zrekonstruować tysiące genomów pochodzących z metagenomu (MAG) bezpośrednio ze zsekwencjonowanych próbek środowiskowych bez potrzeby hodowli drobnoustrojów w laboratorium” – powiedział Stephen Knifach, genetyk i autor badań w Joint Genome Institute przy Departamencie Energii USA.
„To, co naprawdę odróżnia to badanie od poprzednich, to niezwykła różnorodność ekologiczna analizowanych przez nas próbek”.
Zespół miał dostęp do ogromnej bazy danych zawierającej ponad 10 000 metagenomów – termin obejmujący cały materiał genetyczny z próbek środowiskowych. Każde DNA, które mogą wyodrębnić, jest klonowane, a następnie sekwencjonowane przy użyciu maleńkich nici genomu, zanim naukowcy ponownie spróbują połączyć te krótkie fragmenty DNA.
To tak, jakby próbować poskładać układankę, która została połączona, ale przy użyciu techniki zwanej “ binningiem '' zespół był w stanie poskładać 52 515 MAG z danych – wiele z nich jest wysokiej jakości i wszystkie mają ponad 50 procent kompletnego genomu.
To nie pierwszy raz, kiedy naukowcy odkryli mikroby za pomocą metagenomiki – w 2018 roku odkryli 16 gigantycznych wirusów, aw 2017 roku nowymi metodami odkryli 20 nowych ewolucyjnych gałęzi na drzewie życia.
Jednak w ramach tej nowej pracy naukowcy próbowali analizować próbki z wielu różnych obszarów, aby wypełnić niektóre luki w naszej wiedzy o drobnoustrojach.
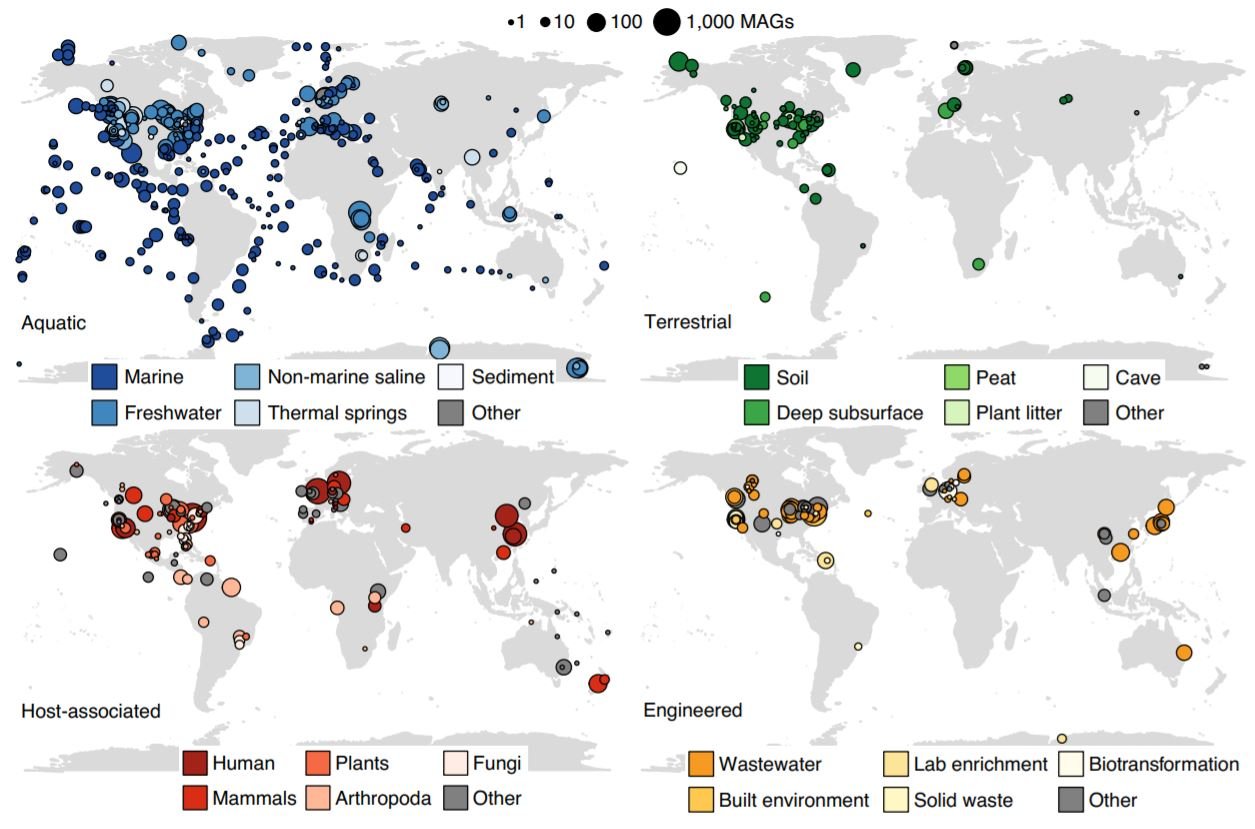
Spójna mapa lokalizacji i typów MAG. (Naifach i in., Nature Biotechnology, 2020).
“Przeprowadziliśmy montaż metagenomiczny i binning na 10450 rozmieszczonych na całym świecie metagenomach z różnych siedlisk, w tym oceanów i innych środowisk wodnych, środowisk związanych z ludźmi i zwierzętami, a także gleb i innych środowisk lądowych, aby przywrócić 52 515 MAG”, genetyka.
„Katalog poszerza znaną różnorodność filogenetyczną bakterii i archeonów o 44 procent”.
Kiedy naukowcy zbadali genomy izolatów, MAG z poprzednich badań i genomy z pojedynczych komórek, odkryli, że 12 556 z 50 000 MAG nigdy wcześniej nie zostało zsekwencjonowanych.
Teraz ważne jest, aby pamiętać, że te genomy nie są tak dobre, jak te, które można uzyskać z hodowli bakterii i archeonów w laboratorium, a następnie ich sekwencjonowania. Proces fuzji może mieszać części genomu między gatunkami bakterii, a fragmentów genomu często brakuje; bez możliwości hodowania takich gatunków w laboratorium, ale wciąż jest to świetny sposób na wykrycie drobnoustrojów w otaczającym nas świecie.
Badanie zostało opublikowane w Nature Biotechnology.
Źródła: Zdjęcie: (Rodolfo Parulan Jr / Getty Images)